A Profa. Dra. Fernanda Machado Fonseca, docente adjunta do Curso de Biomedicina, do Campus da UFPI em Parnaíba, publicou o artigo "MLST-Based Population Genetic Analysis in a Global Context Reveals Clonality amongst Cryptococcus neoformans var. grubii VNI Isolates from HIV Patients in Southeastern Brazil" no renomado periódico internacional PLOS Neglected Tropical Diseases. O artigo conta com a participação de pesquisadores da Austrália, Estados Unidos, Itália, Tailândia, Holanda, Reino Unido além de diversas instituições brasileiras.
O estudo é resultado de uma parceria iniciada pela Prof. Fernanda há cerca de dois anos, quando já mantinha contato com pesquisadores brasileiros da Universidade Federal do Triângulo Mineiro (UFTM) e então teve a oportunidade de desenvolver a pesquisa com o grupo de Micologia Clínica da instituição "The University of Sydney", na Austrália, local onde os experimentos foram desenvolvidos.
No trabalho, foram avaliados isolados clínicos de pacientes HIV positivos e ambientais de Cryptococcus neoformans var. grubii VNI da região sudeste do Brasil por MLST. Os membros do complexo Cryptococcus neoformans / Cryptococcus gattii são a causa da criptococose, uma doença humana potencialmente fatal, responsável por 624 mil mortes por ano. A maioria dos casos de meningite em pacientes infectados pelo HIV é causada por C. neoformans. A infecção é adquirida por inalação de células de levedura desidratadas de fontes ambientais.
Os dados obtidos demonstraram uma estrutura populacional clonal dos isolados, com baixa variabilidade quando comparados com isolados de diversos continentes. Segundo a pesquisadora, esta baixa variabilidade provavelmente se deve a múltiplos eventos de dispersão da África para as Américas. A maioria dos isolados clínicos foi de um tipo de sequência (ST93), que também foi encontrada em amostras ambientais. Ao expandir a análise para isolados de todo o mundo, foi possível identificar dois grupos principais entre C. neoformans var. grubii VNI.
Confira o artigo completo http://journals.plos.org/plosntds/article?id=10.1371/journal.pntd.0005223.
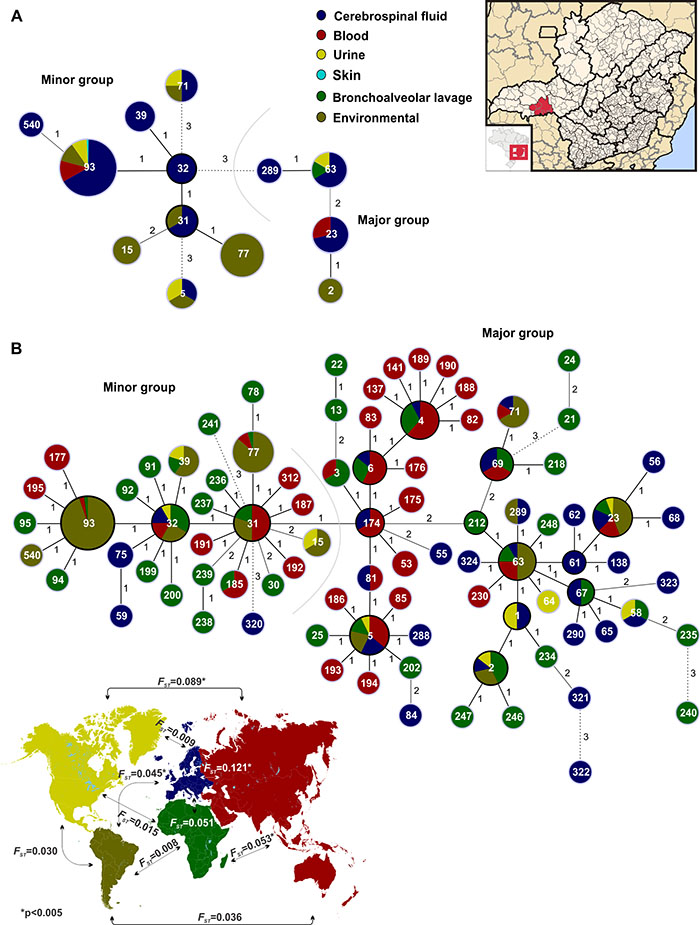
"Distribuição dos isolados de Cryptococcus neoformans provenientes do Brasil (A) e comparados com isolados provenientes de diversos países (B) por meio do sequenciamento de DNA revelando a presença dos dois grupos filogenéticos no genótipo VNI."